近日,国际学术期刊Biosafety and Health在线发表了中国科学院上海营养与健康研究所生物医学大数据中心张国庆研究团队的论文“An explainable artificial intelligence framework reveals mutations associated with drug resistance in Mycobacterium tuberculosis”。研究团队开发了一种名为xAI-MTBDR的可解释人工智能框架,基于近4万株结核分枝杆菌全基因组测序数据,不仅实现了耐药性的高精度预测,还系统揭示了潜在耐药新位点及其可能的分子机制,为结核病的精准诊断与治疗提供了新的技术工具。
结核病是由结核分枝杆菌(Mycobacterium tuberculosis, MTB)引起的全球性细菌传染病,2023年全球约有1060万新发病例并导致130万人死亡。耐药结核病的出现显著降低了治疗成功率,进一步加剧了结核病防控的难度。近年来,机器学习方法已被用于从基因组数据中识别与耐药相关的复杂模式,但现有研究多停留在群体层面的统计关联,缺乏对单个菌株耐药机制的可解释性分析,而这一层面的解析对于理解特定突变如何介导耐药具有关键意义。因此,亟需能够同时提供群体层面“全局解释”和个体层面“局部解释”的预测模型:前者用于系统评估突变对耐药性的整体贡献,后者用于揭示具体菌株耐药表型的关键驱动因素。
基于此,研究团队构建了可解释人工智能模型xAI-MTBDR,通过集成多种机器学习模型与SHAP方法,旨在识别新的耐药相关突变并准确预测MTB的药物耐药性。利用包含39,145株MTB分离株的公共数据集进行评估,xAI-MTBDR在预测所有一线抗结核药物耐药性方面表现优于当前最先进的方法,并能对每个突变在耐药性中的贡献进行量化评分。模型的预测结果与世界卫生组织发布的MTB耐药突变目录高度一致,验证了其广泛的适用性与可靠性。在个体层面,xAI-MTBDR能够解析突变对每一株分离株耐药的贡献。本研究共发现27个潜在的耐药标志物,其中部分突变在蛋白质三维结构中比已知耐药突变更接近对应药物的结合位点,提示其可能在介导耐药性中发挥更直接的作用。此外,基于贡献评分,该框架可对分离株进行有效亚群划分,反映其耐药程度的差异。
综上,xAI-MTBDR为耐药结核分枝杆菌的精准检测提供了有力工具,并为理解耐药机制提供了群体与个体层面的新视角。随着全基因组测序与耐药表型数据的不断积累与完善,整合可解释人工智能方法有望进一步提高预测准确性,深化对耐药机制的理解,优化临床治疗策略,从而为降低耐药结核病及其他病原体感染所致的疾病负担提供支持。
中国科学院上海营养与健康研究所生物医学大数据中心张国庆研究员和赵国屏院士为论文共同通讯作者;博士研究生岑卉、生物医学大数据中心副研究员张鹏和高级工程师凌鋆超为论文共同作者。该研究得到了科技部国家重点研发计划、上海市科技创新行动计划、广州国家实验室研发计划等项目的资助。
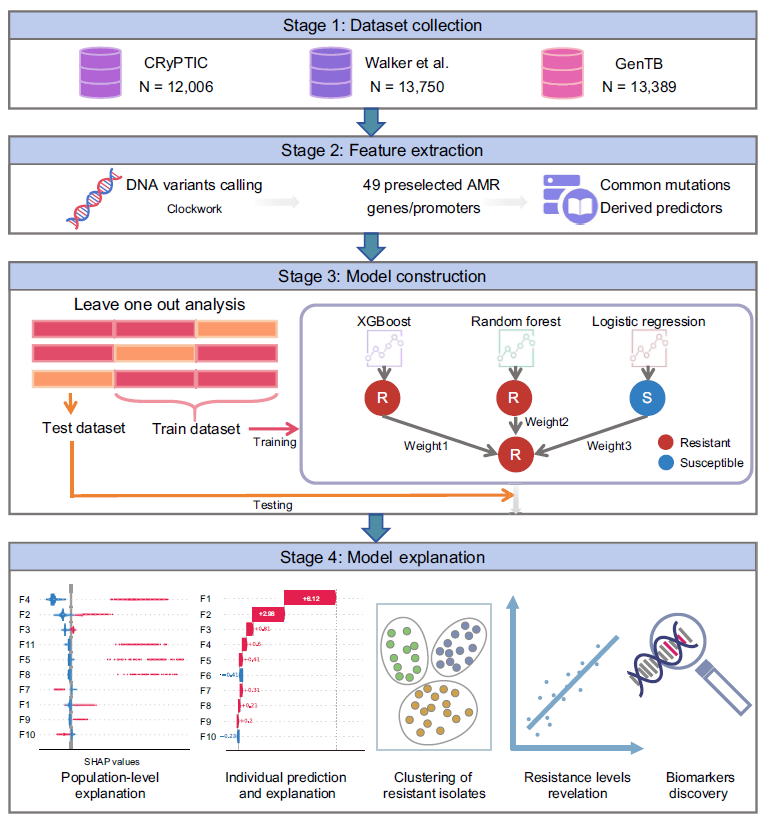
图:可解释性结核耐药分析模型xAI-MTBDR示意图
论文链接:https://doi.org/10.1016/j.bsheal.2025.11.001
推送单元:张国庆研究组、科技规划与任务处